【cytoscape下载】cytoscape中文版 v3.7.2 最新版(附安装和使用教程)
软件介绍
cytoscape是一款专业的信息数据分析和编辑软件,它的主要功能是帮助用户分析各种数据,通过多种形式的网络图形为用户显示分析结果,让用户能够更好地了解数据之间的关联和变化趋势。cytoscape通常被使用在生物信息分析等领域,有需要的用户敬请下载。
特色功能
以多种标准格式加载分子和遗传相互作用数据集
项目和集成全局数据集和功能注释
在这些数据之间建立强大的可视化映射
使用细胞视图应用程序执行高级分析和建模
可视化和分析人为策划的路径数据集,如维基路径、反应堆和KEGG。
安装教程
JAVA安装:java8是否已安装?
如果没有安装或环境变量的配置等,安装Cytoscape时候点击提示出:是否安装java,点击是即可。
其他默认安装就可以,可以更改安装目录。(但不建议瞎改)
安装好了之后需要设置环境变量。
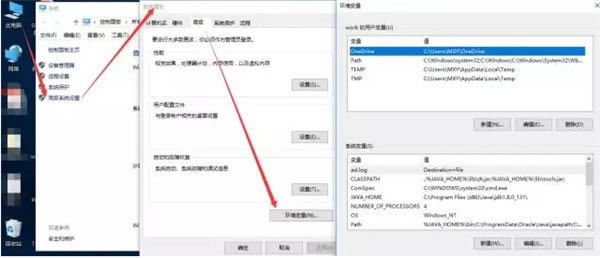
在环境变量中,要修改两个地方,一个是添加JAVA_HOME。选择“新建”,变量名填上JAVA_HOME,变量值填上C:\Program Files\Java\jdk1.8.0_151,在java的安装过程中,默认一直下一步安装,所以装在C盘,如果你在安装过程中改了,那可能是D盘或者E盘,那么变量值要做相应的更改。
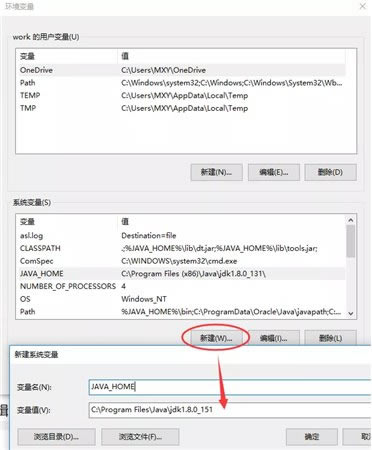
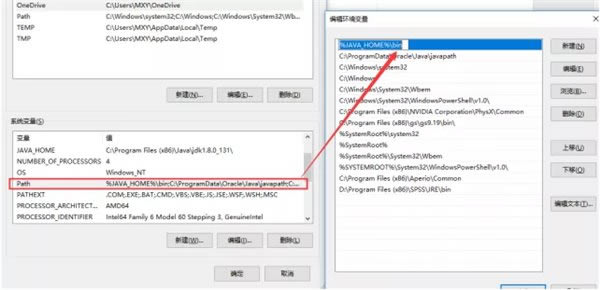
还要修改一个地方,就是Path,添加JAVA的变量值到Path中:选择Path,然后点“编辑”,在最后面添加如下语句:%JAVA_HOME%\bin。
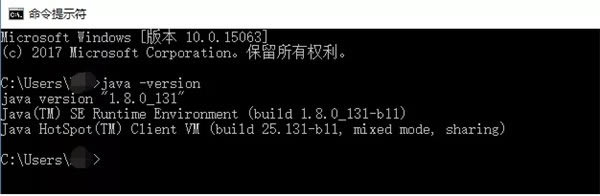
打开命令提示符cmd,输入java -version,如果能正常显示,那表明装好了,你就可以装Cytoscape了。
使用教程
1. 安装并打开Cytoscape
2. 导入蛋白质互作网络数据库中参考物种的蛋白互作网络
网络文件格式包括:TXT、SIF、GML等,在Cytoscape安装目录下有Sample Data,是部分网络文件的示例。
本地导入参考物种的蛋白互作网络
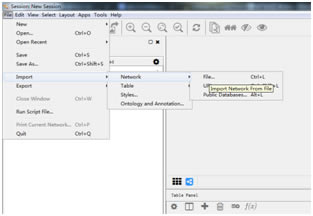
Cytoscape默认将文件第一行作为列名。如果文件第一行是基因,在导入网络时要在Advanced Options中取消用第一行作为列名。
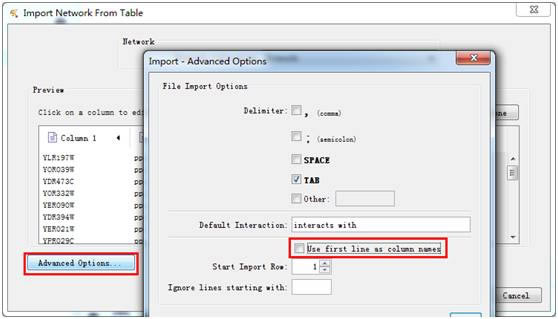
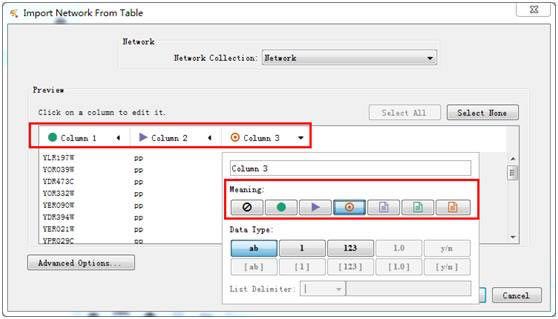
然后选择每一列数据的属性,包括Source Node、Interaction Type、Target Node、Edge Attribution、Source Attribution、Target Attribution等,同时对应不同颜色和图标标记。
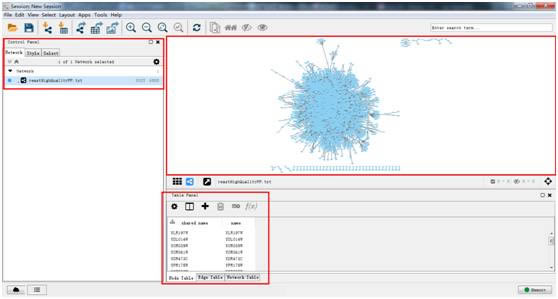
导入后,右上部窗口内展示蛋白互作网络图,右下部为网络图的相关节点 (Node) 及边 (Edge) 的数据信息。左侧为导入的网络目录和网络属性 (Style) 设置菜单。
从公共数据库中获得参考物种的蛋白互作网络
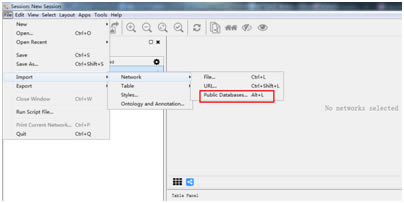
例如要导入某个物种的蛋白质互作网络:选择物种和想要查看的数据库,搜索包含该物种蛋白互作网络的数据库,导入数据(时间稍长)。
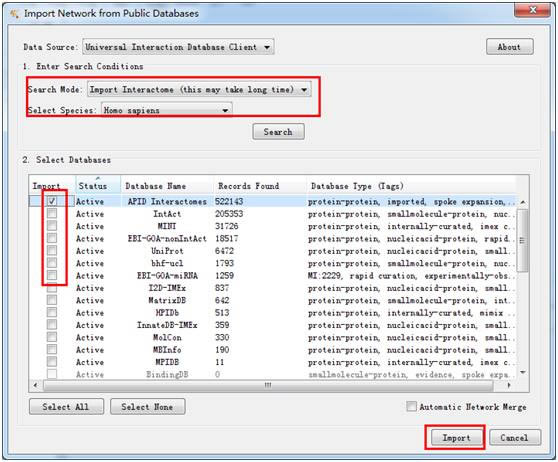
3. 在Layout选项下可以选择方便观测的布局(默认导入的布局方式为Perfuse Force Directed Layout)。
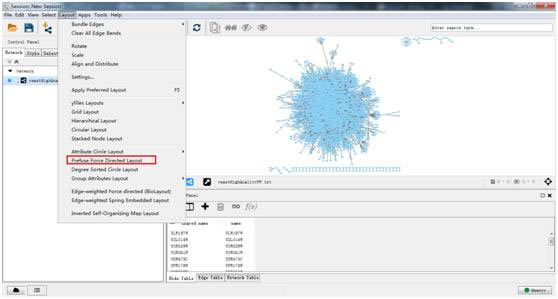
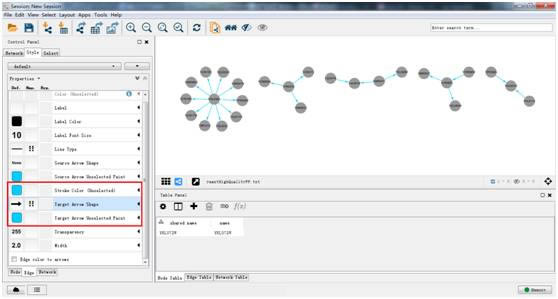
4. 设置网络节点和边的风格 (Style)
在Style选项卡下Node设置中对点的形状、颜色、大小等进行设置。例如:选择Shape为圆形、Fill Color为灰色、节点的高度 (Meight) 和宽度 (Width) 为50.0。
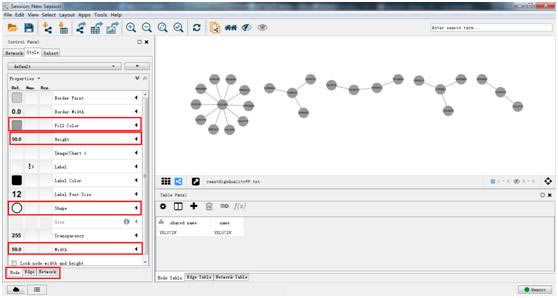
在Style选项卡下Edge设置中对边的形状、颜色、大小等进行设置。例如,将连线的颜色设置为蓝色,同时设置到Target方向的箭头同样为蓝色。
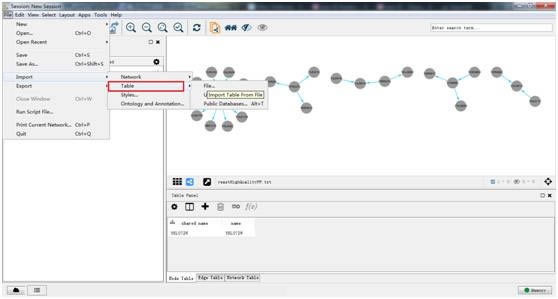
5. 导入差异表达基因及其属性列,映射到互作网络中作为各节点的拓扑性质。
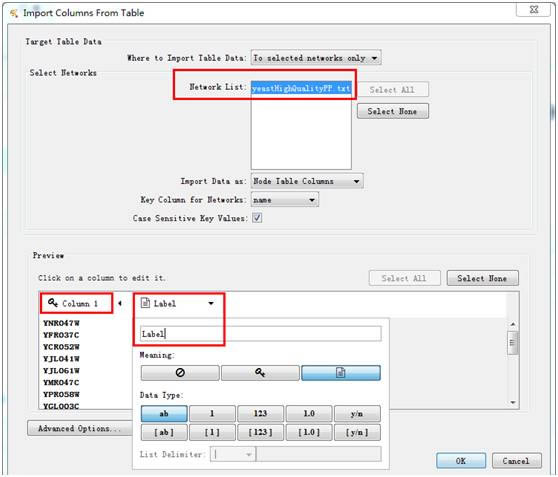
选取目标网络、设置关键列 (Column1)、选择需要的节点属性列(可以选取多列,本例选取Column2,并重命名为Label,其中为up/down数据)。
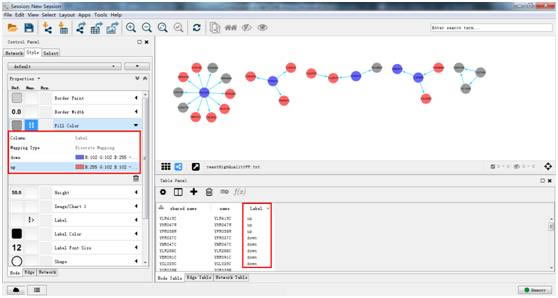
6. 根据上下调基因 (up/down) 标示网络,定义上调基因 (up) 为红色,下调基因 (down) 为蓝色。
7. 导出数据
导出图像Export Network as Graphics。
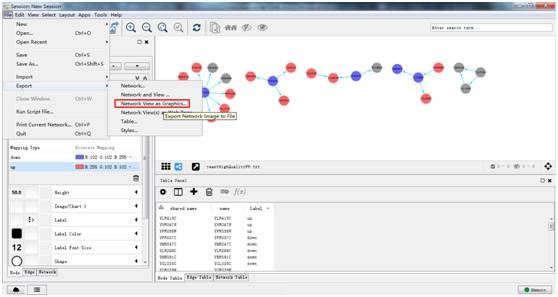
导出数据 Export Table。
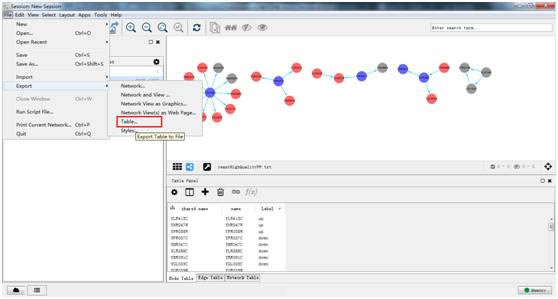
可导出数据面板中的网络节点、边和整体网络的信息。
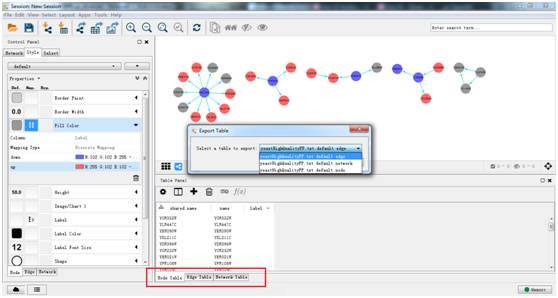
在弹出窗口编辑导出文件名。
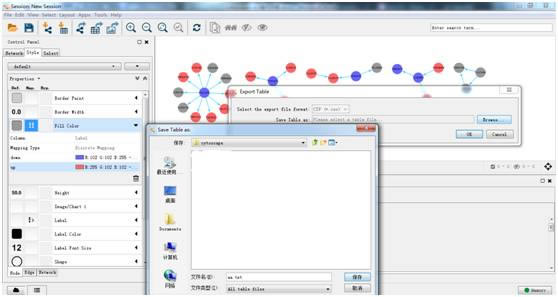
……
点击下载行业软件 >>本地高速下载
点击下载行业软件 >>百度网盘下载